I was visiting my friend Miroslaw Kwasniewski an Assistant Professor at the University of Silesia in Katowice, Poland. He is got a GS Junior 454 machine which I wanted to check out. Besides that, he wanted some help with their bioinformatics servers and pipelines.
I was just looking at one dataset, transcriptomics from barley, obtained from the GS Junior, and read counts, read lengths and quality are all great, very impressive. I have some statistics obtained using FastQC, after exporting the SFF file from GS Junior to FastQ format using sff_extract.
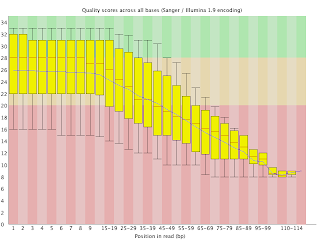
This is the distribution of the read quality on a per base basis. As you can see, reads can be well over 400, around 500bp with Phred qualities above 20.
The sequence length distribution has a nice peak around 500 bp.
GS Junior could achieve 100.000 reads throughput, Mirek was getting 150.000 reads.
GS Junior seem to be quite good for sequencing low complexity samples. Quality and length of the sequencing reads is great, but depth for a e.g., full transcriptome in angiosperms, would be either challenging or too expensive. It is great for amplicon sequencing, bacterial genome sequencing, virus genome sequencing, i.e., would be great for the phages (!).
martes, junio 07, 2011
viernes, junio 03, 2011
Secuenciación del genoma de cepa de E. coli que está causando problemas en Alemania
En mayo de 2011 un brote de diarrea hemorragica se desató en los estados de la región norte de Alemania. Varios de los casos reportados causaban sindrome urémico hemolítico, caracterizado por insuficiencia renal y transtornos neurológicos. Investigadores alemanes identificaron que el agente responsable de brote es una nueva variante de la bacteria Escherichia coli entero-hemorrágica (EHEC, por sus siglas en inglés) serotipo O104.
El Instituto de Genómica de Beijing (BGI) acaba de liberar las secuencias crudas obtenidas usando Ion Torrent de la cepa de E. coli que está causando el brote de EHEC en Alemania.
El BGI liberó las lecturas crudas correspondientes a 5 corridas del Personal Genome Machine (PGM) de Ion Torrent, los datos están disponibles en el NCBI SRA con el número de acceso: SRA037315. Los datos se pueden descargar en formato .sra. El SRA tiene un kit de software que permite convertir el formato sra en fastq o sff. Yo los exporte en formato fastq, usando el comando:
La opción tal vez mas importante en este comando es -W que remueve los extremos de baja calidad de las lecturas.
Uno de los primeros paso antes de continuar al ensamble de las lecturas es revisarla un poco, e.g., longitud, calidades. Este tipo de análisis se puede adelantar con FastQC.
La calidad es buena hasta la posición ~40, a partir de allí cae drasticamente. No muy sorprendente para este tipo de tecnologías. Las gráficas con similares para las otras 4 corridas. La siguiente figura corresponde a la distribución de calidad en todas las corridas (archivos fastq concatenados).
Sobre la longitud de las secuencias, la Figura de abajo, muestra que el PGM genera lecturas en un rango bastante estrecho, con un pico en 100bp.
El Instituto de Genómica de Beijing (BGI) acaba de liberar las secuencias crudas obtenidas usando Ion Torrent de la cepa de E. coli que está causando el brote de EHEC en Alemania.
El BGI liberó las lecturas crudas correspondientes a 5 corridas del Personal Genome Machine (PGM) de Ion Torrent, los datos están disponibles en el NCBI SRA con el número de acceso: SRA037315. Los datos se pueden descargar en formato .sra. El SRA tiene un kit de software que permite convertir el formato sra en fastq o sff. Yo los exporte en formato fastq, usando el comando:
for i in $(ls *.sra); do name=`basename $i .sra`; fastq-dump -TR -W -F -SL -A $name $i; done
La opción tal vez mas importante en este comando es -W que remueve los extremos de baja calidad de las lecturas.
Uno de los primeros paso antes de continuar al ensamble de las lecturas es revisarla un poco, e.g., longitud, calidades. Este tipo de análisis se puede adelantar con FastQC.
| Corrida (SRA accession) | Número de Lecturas | Rango de tamaños |
|---|---|---|
| SRR227300 | 92370 | 1-119 |
| SRR227337 | 122208 | 1-129 |
| SRR227338 | 96765 | 1-125 |
| SRR227339 | 222275 | 1-131 |
| SRR227340 | 95750 | 1-129 |
La calidad es buena hasta la posición ~40, a partir de allí cae drasticamente. No muy sorprendente para este tipo de tecnologías. Las gráficas con similares para las otras 4 corridas. La siguiente figura corresponde a la distribución de calidad en todas las corridas (archivos fastq concatenados).
Sobre la longitud de las secuencias, la Figura de abajo, muestra que el PGM genera lecturas en un rango bastante estrecho, con un pico en 100bp.
Como podemos observar los datos generados por el PGM de Ion Torrent son lecturas de ADN de longitud corta. En general este tipo de datos se prestan muy bien para hacer ensambles de mapeo, pero no muy bien para ensambles de novo. A pesar de estoy yo estoy interesado en ver las posibilidades y las limitaciones de este tipo de datos para los ensambles de novo. Mas adelante les contaré los resultados de estos experimentos.
Suscribirse a:
Entradas (Atom)